유전 연구를 수행하는 과정에서 우리는 종종 작은 해부학적 구강 종양, 심지어 단일 세포 샘플 및 인간 세포에서 매우 낮은 수준으로 전사되는 특정 유전자 돌연변이 샘플을 연구하기 위한 RNA 샘플이 부족한 경우가 있습니다.물론 COVID-19 테스트의 경우 샘플링 중에 면봉이 올바른 위치에 있지 않거나 시간이 충분하지 않으면 샘플 크기가 매우 작을 것입니다. 그래서 보건 가족 계획위원회는 이틀 전에 나왔고 테스트를 통과했으며 핵산 샘플러가 6개의 샘플을 채취하지 않은 경우 신고할 수 있습니다.
시약의 민감도가 중요한데 이런저런 문제가 있어서 RT-PCR의 민감도를 높이려면 어떻게 해야 할까요?
가능한 해결책을 논의하기 전에 방금 언급한 상황과 관련된 두 가지 큰 문제를 언급하겠습니다.
우선 샘플에 세포 집단이 몇 개밖에 없을 때 RNA 손실에 대해 걱정합니다.컬럼 방식이나 핵산 침전 방식과 같은 기존의 분리 세척 방식을 사용하면 소수의 샘플이 손실될 가능성이 높습니다.한 가지 해결책은 tRNA와 같은 캐리어 분자를 추가하는 것이지만, 그 경우에도 우리의 복구 실험이 OK라는 보장은 없습니다.
그래서 더 좋은 방법은 무엇입니까?배양 세포 또는 미세해부학적 샘플에 대한 좋은 옵션은 직접 용해를 사용하는 것입니다.
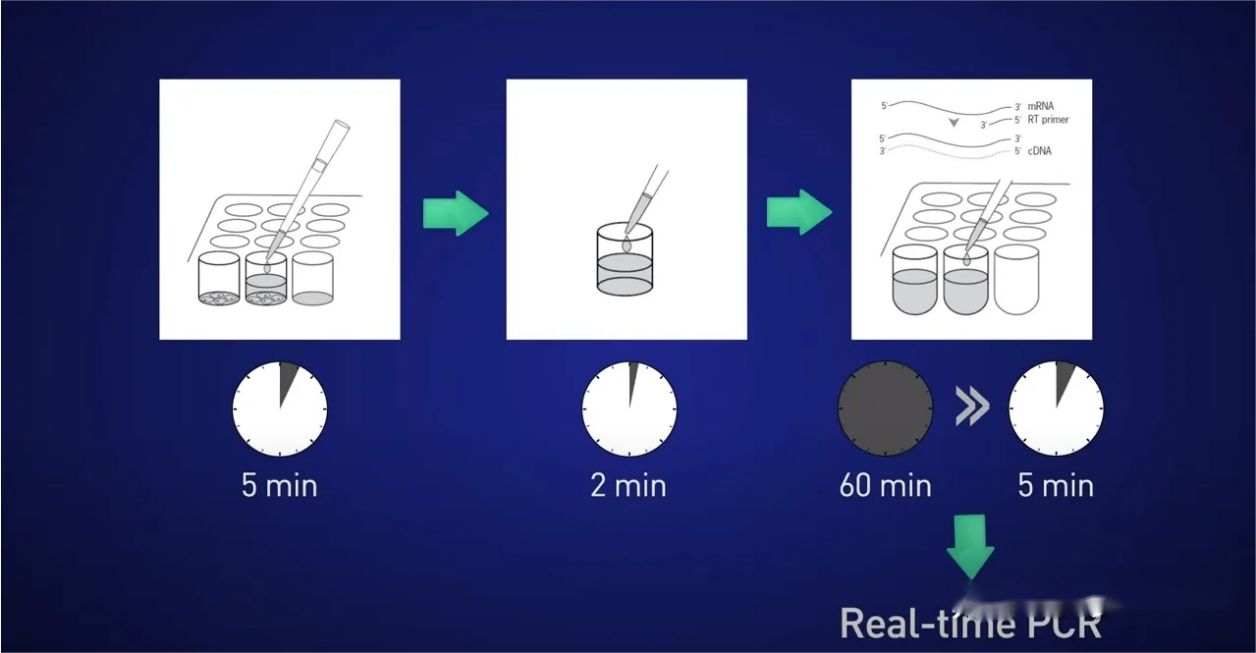
아이디어는 5분 동안 세포를 분할하고 RNA를 용액으로 방출한 다음 2분 동안 반응을 중지한 다음 RNA가 손실되지 않도록 lysate를 직접 역전사 반응에 추가하고 최종적으로 생성된 cDNA를 직접 넣는 것입니다. 실시간 반응으로.
그러나 제한된 시작점이나 적은 양의 표적 유전자 발현으로 인해 모든 RNA를 재활용할 수 있지만 좋은 실시간 신호를 얻기에 충분한 템플릿을 제공하지 못한다면 어떻게 될까요?
이 경우 사전 증폭 단계가 매우 유용할 수 있습니다.
다음은 역전사 후 민감도를 높이기 위한 방안이다.시작하기 전에 사전 증폭을 위해 이러한 표적에 대한 특정 프라이머를 설계하기 위해 어떤 표적에 관심이 있는지 다운스트림에 문의해야 합니다.
이는 최대 100쌍의 프라이머와 10~14회의 반응 주기로 혼합된 프라이머를 생성함으로써 달성할 수 있습니다.따라서 이 요구 사항에 맞게 특별히 설계된 마스터 믹스는 얻은 cDNA를 사전 증폭하는 데 필요합니다.
사이클 수를 10~14 사이로 설정한 이유는 이 제한된 사이클 수로 인해 다양한 표적 사이의 무작위성이 보장되기 때문에 정량적 분자 정보가 필요한 연구자에게 매우 중요합니다.
pre-amplification 후 많은 양의 cDNA를 얻을 수 있어 back-end에서의 detection 감도가 크게 향상되고 샘플까지 희석하여 여러 번의 real-time PCR 반응을 수행하여 가능한 random error를 제거할 수 있습니다.
게시 시간: 2023년 4월 11일